KAPA Unique Dual-Indexed Adapter Kit
KAPA Unique Dual-Indexed Adapter Kit
(0件)
よくあるご質問4件
Illumina社シーケンサー用のライブラリー調製に使用するためのアダプター溶液で構成されたキット。DNAおよびRNAシーケンシングアプリケーション用のKAPAライブラリー調製キットと組み合わせて使用する事が可能
特長
- KAPAが開発した独自配列のユニーク・デュアルインデックスアダプター
- 従来のDual Indexアダプターでは解決できなかった「インデックスホッピング」に対応可能
- illumina社Experiment Managerの設定資料あり
- 整列化フローセル装置でのインデックス・ミスアサインメントの抑制に効果的なユニークインデックス
- NGSでのQCを実施した高品質アダプター
- 推奨キット:KAPA HyperPrep、KAPA HyperPlus、KAPA RNA HyperPrep および HyperCap workflows
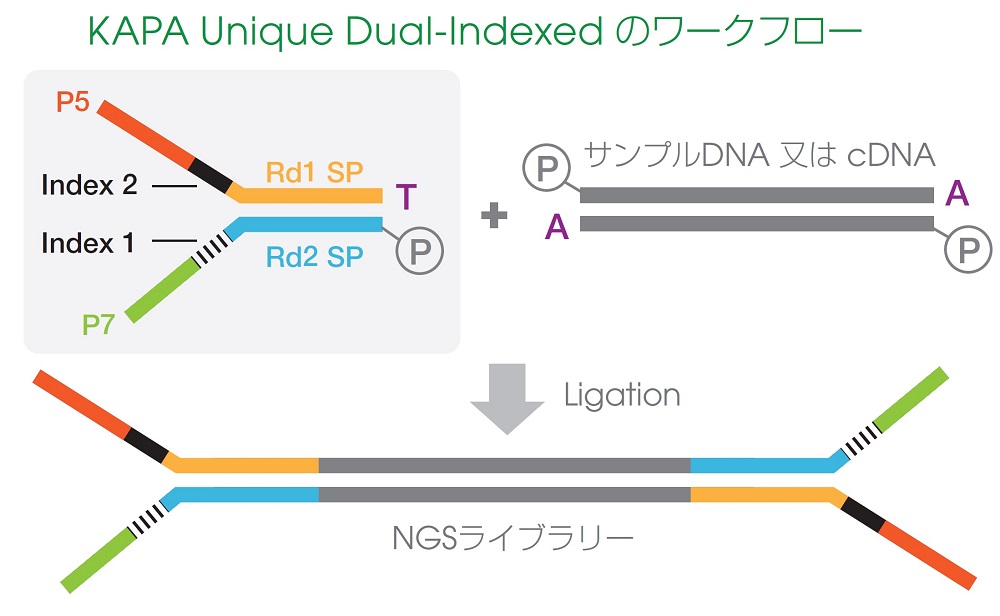
適用システムおよびアプリケーション
本キットはIllumina社のシーケンサーによる以下のアプリケーション用のライブラリー調製に使用することができます。
- ヒト全ゲノムシーケンシング(human WGS)
- ハイスループットのエクソーム解析、ターゲットシーケンス、RNAシーケンス
- 微生物全ゲノムシーケンシングおよびその他のショットガンシーケンシング
本キットに含まれるアダプター配列はメチル化されていませんので、Methyl-seq(バイサルファイト処理)に使用する事はできません。
製品構成
本キットはIllumina 社のシーケンサーに対応したアダプター溶液と、濃度調整のためのアダプター希釈バッファー、予備のピアス可能なシーリングフィルム3枚で構成されています。
濃度は15 µMで、各アダプター溶液は20 µLずつ、96ウェルプレートに分注されていますが、インプット核酸の量や断片長、ライブラリー調製キットの種類に応じて適切な濃度のキットを選択し、また、適宜アダプター希釈バッファーで希釈してからご使用ください。

セレクションガイド
ライブラリー調製キットの種類や初発核酸の使用量によりどちらのキットを使用するべきかをご選択ください。
| KAPA Unique Dual-Indexed Adapter | KAPA Dual-Indexed Adapter | |
|---|---|---|
| インデックス数 | 96 | |
| デュアルインデックス配列 | ユニーク | 組み合わせ型 |
| 8塩基のP5バーコード種類 | 96 | 8 |
| 8塩基のp7バーコード種類 | 96 | 12 |
| バーコード配列 | KAPAオリジナル *1 | illumina社同様 |
| インデックスミスアサインメント(インデックスホッピング)対応 | Yes | No |
| 整列化フローセル装置での使用 (例:HiSeq®3000,HiSeq 4000,HiSeq X,NovaSeq™) |
Yes | No |
| 従来型フローセル装置での使用 (例:Miseq™,NextSeg™,HiSeq 2000,HiSeq 2500) |
Yes | |
| KAPA HyperPrep、KAPA HyperPlus、KAPA RNA HyperPrepおよびHyperCap workflowsとの組み合わせ | Yes | |
| Roche Universal Blocking Oligosとの組み合わせ | Yes | |
| Methyl-seq(バイサルファイト処理)での利用 | No *2 | |
| アダプター溶液の仕様 | 濃度:15 µM ; 液量:20 µL /各アダプター *3 | |
| Kit 仕様 | ハードシェルプレートに分注済み、交換用シール入り *4 | |
| KAPA Adapter 希釈バッファー | 付属 (25 mL) | |
*1 KAPA独自の192バーコード配列を使用。
*2 Methyl-SeqはSeq Cap Adapter Kitをご利用ください。
*3 アダプターを希釈しない場合、各インデックスがKAPA HyperPrepまたはKAPA HyperPlusで4回分のライブラリー作製にご使用いただけます。
*4 アダプター容器のプレートは剥離可能なシールで密閉して出荷されます。
交換用のシールはユニークデュアルインデックスアダプターとデュアルインデックスアダプターで仕様が異なります。
- ユニークデュアルインデックスアダプター:ピアスと剥離の両方が可能なシール
- デュアルインデックスアダプター:剥離が可能なシール
キット内容
- 15 µMの調整済み溶液(96ウェルプレートに、20 µLずつ分注済み)
- アダプター希釈バッファー 25 mL
- ピアス・剥離可能なシーリングフォイル 3枚




