本パネルはKAPA HyperCap Design Share Panel(プレデザインパネル)の一つで、非ホジキンリンパ腫(NHL)の血中ctDNAを次世代シーケンサーにより高感度に検出し、経時的な解析を行うための研究用パネルです。
非ホジキンリンパ腫 (NHL)とは?
非ホジキンリンパ腫 (NHL) は最も一般的な血液悪性腫瘍の 1 つであり、 2020 年には世界中で 544,000 人の新規症例と
260,000 人の死亡を引き起こしたと推定されています。 *1 *2
リンパ系腫瘍の診断方法には様々なものがありますが、治療中および治療後のモニタリングも重要となってきており、
非ホジキンリンパ腫の状態をモニタリングする上でcfDNA/ctDNAは重要なバイオマーカーとして知られるようになりました*3*4
特長
- 本パネルはNHLに関連する383遺伝子(遺伝子全体または一部)と追加の遺伝子間領域の合計341 Kbをカバーしています。
- ハイブリダイゼーションをベースとしたKAPA HyperCap ワークフロー*5と、オープンソースのバイオインフォマティクス解析ツール*6を
組み合わせて使用します。遺伝子変異の高感度検出と経時的解析を目的とするため、主にSNVを検出するようデザインされています。
高感度なctDNAモニタリングを自施設で
- 新しいNHL臨床研究を開始可能
- さまざまなNHLサブタイプに
- 柔軟な対応(改変)とデータの蓄積でNHL研究を前進
大規模研究*7で実証された独自のパネルデザイン
- 学術研究者やロシュの科学者による10年以上にわたる研究開発を踏襲した実験系*5
- 1,000以上の検体での解析実績*7
- proof of principleデータ *5,*6からのインサイト
シンプルで信頼性の高いワークフロー
- 堅牢なKAPA HyperCap ワークフロー*5を使用
- 研究ニーズに応じてカスタマイズ可能なオープンソースの解析パイプライン*6
- 自動化に適したワークフローでスケールアップが容易
ワークフロー
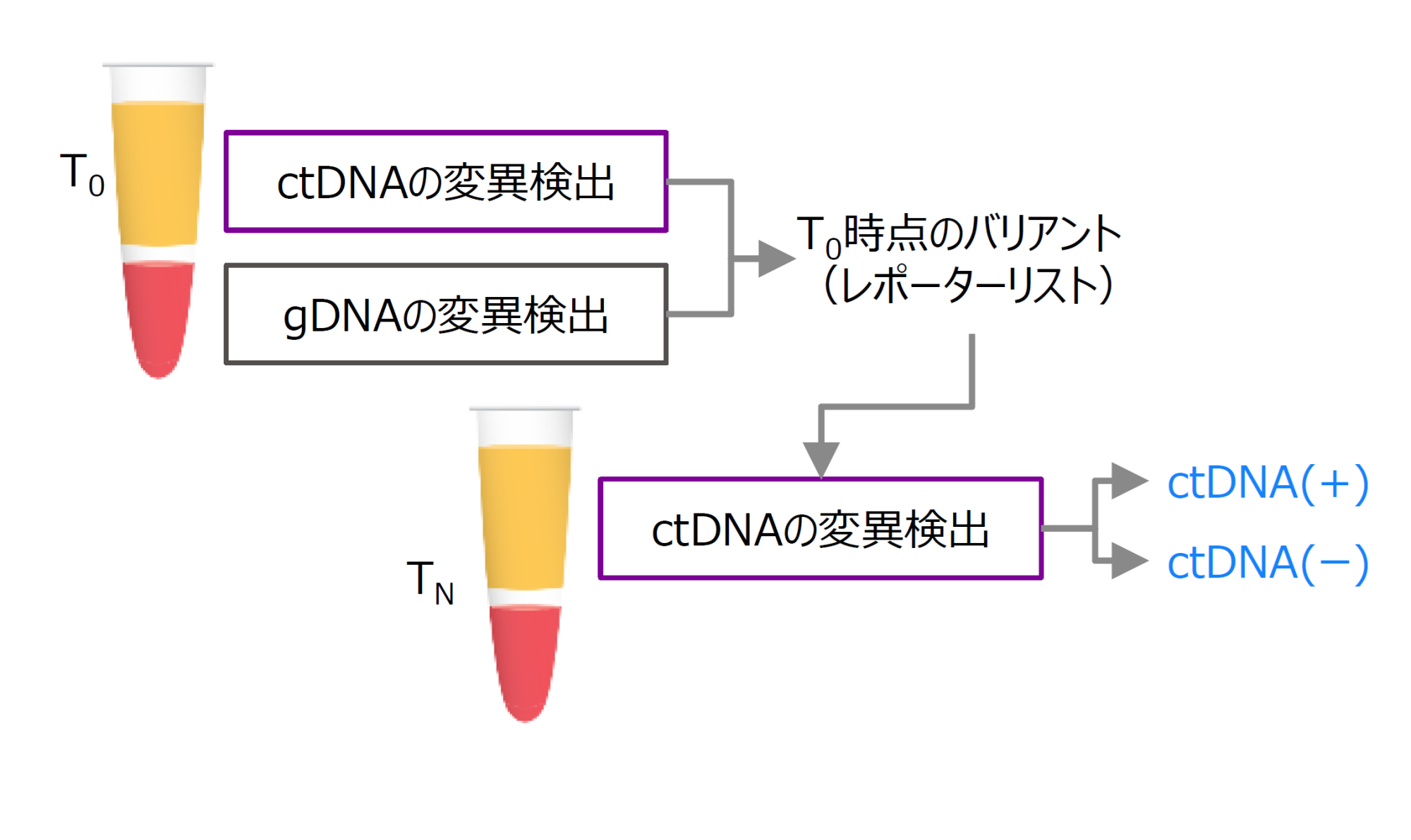
図 1. 微小残存病変(MRD)解析模式図
MRD解析は3つのステップで構成されます。まず健常者のcfDNA検体を用いてSNVブロックリストを作成します。
次に、ベースライン検体(T0)からレポーターリスト作成します(血漿検体と血漿除去後検体での差分とSNVブロックリストの除外)。
最後に、フォローアップ検体(TN)の血漿検体でctDNA陽性または陰性を判定します。
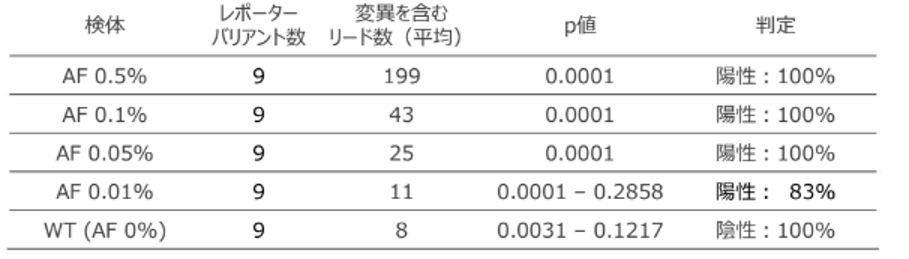
表1. 人工的に調製したcfDNAリファレンス検体を用いた経時的な変異検出解析結果
レポーターバリアントは全てのTN検体で検出されましたが、AFが低いほどサポートリードがゼロではないレポーターバリアントの数は減少し、
また、変異を含むリード数も減少しました。
TN検体でのctDNA陽性/陰性を判定するためのモンテカルロ p値*8,*9のしきい値は、AF 0%検体で観測された最も低い値である0.003に設定しました。
AF0.5%、AF0.1%、AF0.05%では全ての反復実験でctDNA陽性と正確に判定されました。
AF0.01%では、6回の反復実験のうち5回でctDNA陽性と判定されました。AF 0%検体では全ての反復実験で陰性と判断されました。
信頼性の高いKAPA HyperCapワークフロー*7の活用
KAPA NHLパネルでは血漿 (Plasma cfDNA ワークフロー) と 血漿除去後検体( gDNA ワークフロー)の両方を使用します。
実験操作手順や必要試薬の詳細はLongitudinal detection of non-Hodgkin lymphoma ctDNA white paper* 5をご参照ください。
■NHL 血漿ワークフローと必要試薬:KAPA HyperCap cfDNA Workflow v1.1
| 品番 | 品名 |
|---|---|
| 07247737190 | コバス DNAサンプル プレパレーションキット(cfDNA) |
| 09217193001 | KAPA NGS FFPE DNA QC Kit ※ |
| KK8502(7962347001) | KAPA Hyper Prep Kit |
| KK8007(8963835001) | KAPA HyperPure Beads (5 mL) |
| 9329838001 | KAPA UDI Primer Mixes, 97-192 |
| 9329862001 | KAPA Universal UMI Adapter, 960 µL |
| 09075780001 | KAPA HyperCapture Bead kit |
| 09052593001 | KAPA HyperChoice MAX 3Mb T1(IRN:10000028225) |
| 09075810001 | KAPA HyperCapture Reagent kit |
| 09075879001 | KAPA Probes resuspension buffer |
※KAPA NGS FFPE DNA QC Kitに含まれるプライマーは使用せず、下記プライマーをご自身でご用意ください。
・forward primers 66F: 5'-TTGCGGAAGTCAGTGTGG-3'
・forward primers 330F: 5'-CAAACAACCCCATCAAAAAGTG-3'
・reverse primer, 5'-GATGGCTGGGTCAAATGGTA-3'
■NHL 血漿除去後(gDNA)ワークフローと必要試薬:KAPA HyperCap Workflow v3.5
| 品番 | 品名 |
|---|---|
| 09189823001 | KAPA NGS DNA Extraction Kit |
| KK8512(7962401001) | KAPA HyperPlus Library Preparation Kit |
| KK8502(7962347001) | KAPA Hyper Prep Kit |
| KK8007(8963835001) | KAPA HyperPure Beads (5 mL) |
| 9329838001 | KAPA UDI Primer Mixes, 97-192 |
| 9329862001 | KAPA Universal UMI Adapter, 960 µL |
| 09075780001 | KAPA HyperCapture Bead kit |
| 09052593001 | KAPA HyperChoice MAX 3Mb T1(IRN:10000028225) |
| 09075810001 | KAPA HyperCapture Reagent kit |
| 09075879001 | KAPA Probes resuspension buffer |
パフォーマンスデータ
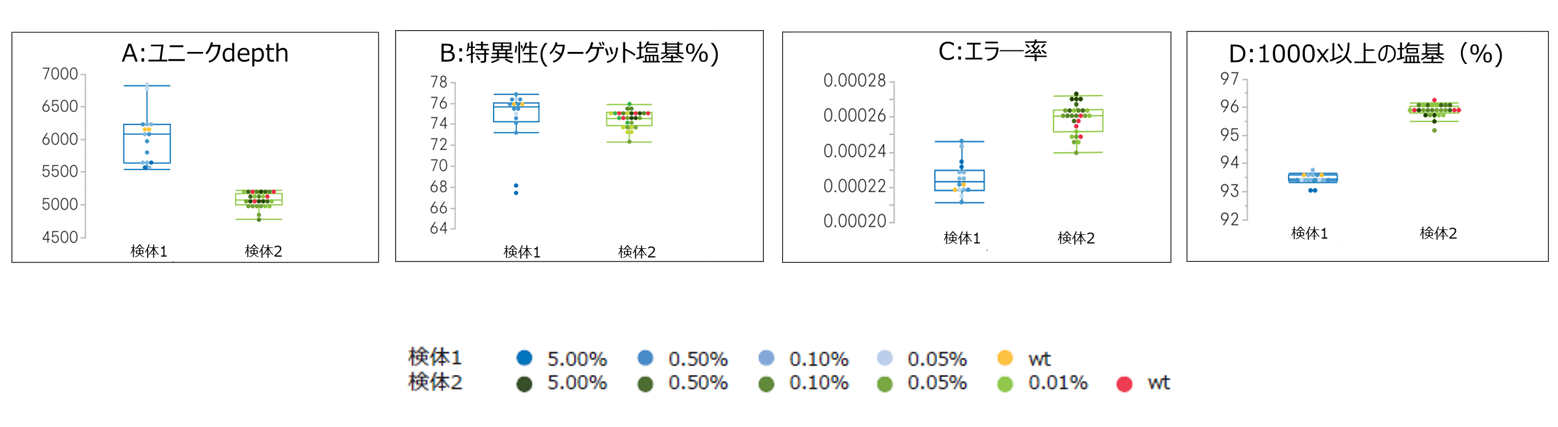
高いシーケンス品質
- 十分なユニークdepth(5,000x以上:図2A):高感度なctDNA検出
- 高い特異性(全データの74%以上がターゲット塩基:図2B):効率の良い解析
- 低いエラー率(約2.5x10-4:図2C):結果への信頼性
図2. KAPA NHLパネルのシーケンシングメトリック
模擬NHL検体として、0%から5%のアレル頻度(AF)のバリアントを含むように市販のcfDNAリファレンス検体(検体1、検体2)を人工的に調製し、
KAPA NHL Plasma cfDNAワークフローの手順に従いNGSライブラリー作成及びエンリッチメントを行いました*5。
イルミナ社のNextSeqTM 500/550の1ランで8検体のシーケンシングを実施して1検体あたり平均8,800万以上のリードを取得し、
オープンソースのバイオインフォマティクスツールを使用してデータ解析を行いました*6。
Unique molecular identifiers(UMI)による重複分子の除去後のメディアン解析リード数は、検体1で44M、検体2では33Mでした。
これはそれぞれ6,100x及び5,000xのユニークdepth(メディアン値)に相当しました。
高い再現性で主要な性能メトリックを十分に満たすことが示され、 KAPA NHLパネルにより効率良くシーケンスできることを示唆しています。
信頼できるバリアントコール
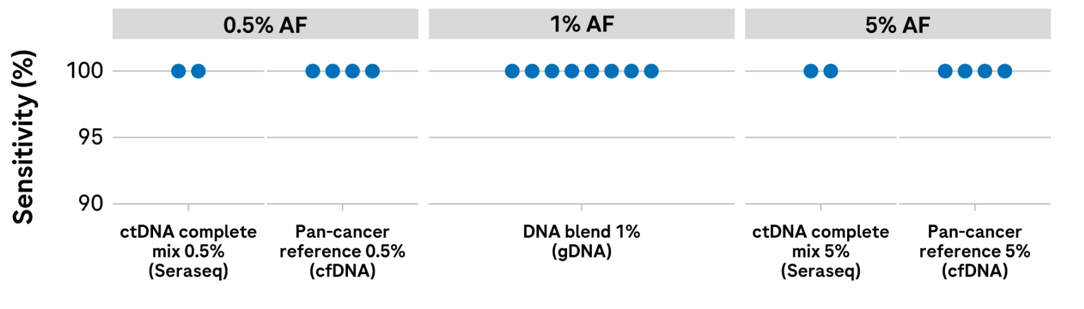
- 0.5% AFのバリアントも高感度で再現性良く検出(図3)
- ⾼い特異性でのバリアントコール*
図3. 0.5%、1%、5% AFのバリアントを再現性良く高感度に検出
人工的に調製した検体で感度と再現性の検証を行いました。ジャームラインバリアントの検出感度を評価するために、NA24631(98%)と
NA24149(2%)のゲノムDNAを混合して1% AFを調製し、KAPA NHL gDNAワークフローを実施しました。青いドットは反復実験での結果を示しています。
その結果、1% AF(真陽性のSNV)10か所全てを8回の反復実験のいずれにおいても検出しました。
ctDNAバリアントの検出感度の評価として、市販のcfDNAリファレンス検体(検体1、検体2)で0.5%または5%AFを調製し、
KAPA NHL Plasma cfDNAワークフローを実施して検証しました。
この結果、予想されるSNV(検体1ではSNV3か所、検体2ではSNV12か所)の全てが、全ての反復実験(検体1は2回、検体2は4回)で検出されました。
*KAPA NHLパネルが高い特異性でバリアントコールできることは次の検証により示されました。
NA24149とNA24631の混合ゲノムDNAを用いた検証実験において、8回の反復実験全てで、53か所の全てが真陰性(TN)と判定されました。
また、変異を持たないと仮定する23名の健康なドナー検体を用いた検証実験では、10Kbあたり0.06個というレベルでのみバリアントが検出され、
バリアントコールのエラー率が非常に低いことが示されました。
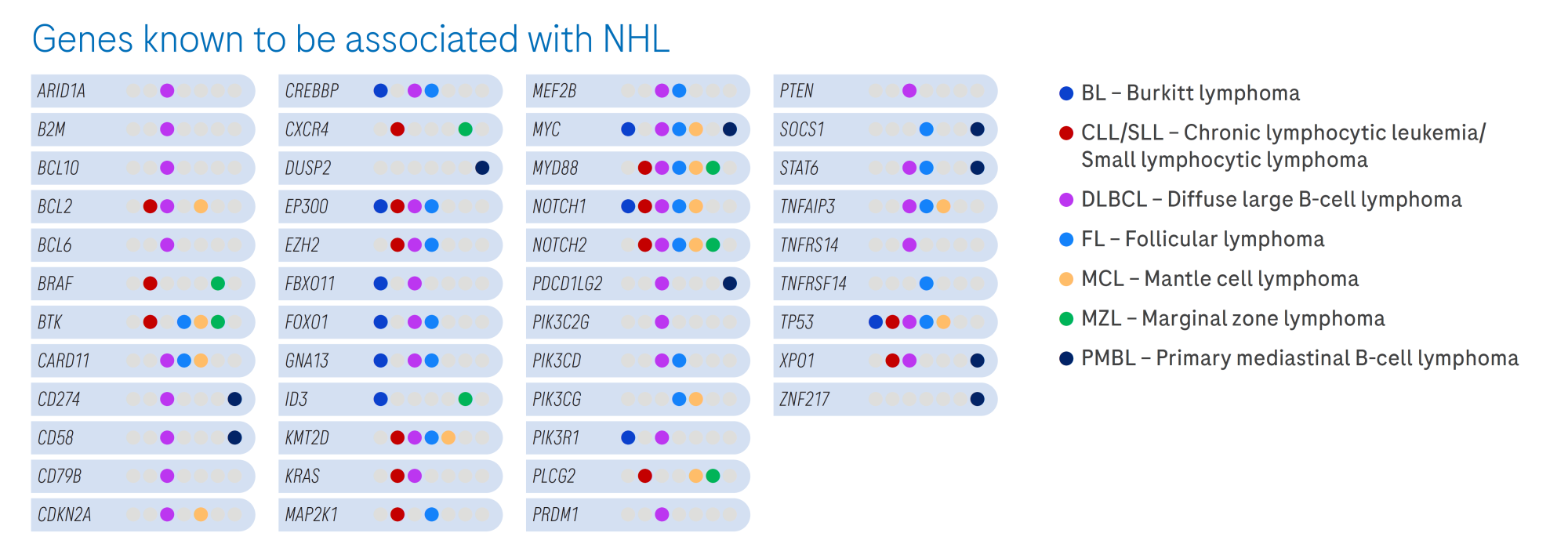
ターゲット遺伝子
KAPA HyperCap DS NHL パネルの総キャプチャーサイズは 341 Kb 。
383遺伝子(完全または一部カバー)に加えて、追加の遺伝子間領域もターゲットとしています。
- 専門家が選択した遺伝子や領域を1つのパネルにすることで、複数のNHLサブタイプに対応
- ctDNA検出、MRD解析、細胞起源決定のような様々な研究に応用可能
- 予後バイオマーカーとしてのctDNAを検証した第3相POLARIX研究*7で使用されたパネルデザイン
すべてのgene listは>>こちら
仕様
- ターゲットサイズ:341 kb
- ターゲット遺伝子:383遺伝子(完全または一部)
- 必要検体:全血:血漿+血漿除去後検体(各30 ng cfDNA+100 ng gDNA)
- 保存温度:受領後–15℃~–25℃/溶解後–15℃~–25℃(※1)
- 使用期限は、凍結乾燥または再懸濁の状態で、ラベル記載に準ずる(製造より36ヵ月)
- プローブデザインファイルはドキュメントタブにある「技術資料」からダウンロードをお願いいたします。
※1
・KAPA Target Enrichement Probesは凍結乾燥された状態で提供されます。
・再懸濁にはKAPA Probes Resuspension Bufferをご使用ください。
・溶解/再懸濁後にはプローブを1回ごとの使用量に分注し、-15℃~-25℃で凍結することをお勧めします。
リファレンス
*1. https://seer.cancer.gov/statfacts/html/all.html. Accessed 22 June 2023
*2. Lou J. Craver A. Bahl K. et al. Etiology of non-Hodgkin lymphoma:A review from epidemiologic studies. JNCC 2022:2(4).226-234.
doi:10.1016/j.jncc.2022.08.003.
*3.Scherer F, Kurtz, DM, Newman AM, et al. Distinct biological subtypes and patterns of genome evolution in lymphoma revealed by circulating tumor DNA.
Sci Transl Med 2016;8(364),364ra155.
doi: 10.1126/scitranslmed.aai8545.
*4.Fernández-Miranda I, Pedrosa L, Llanos M, et al. Monitoring of Circulating Tumor DNA Predicts Response to Treatment and Early Progression in
Follicular Lymphoma: Results of a Prospective Pilot Study. Clin Cancer Res 2023;29(209 – 220).
doi: 10.1158/1078-0432.CCR-22-1654.
*5. Bermejo C, Agarwal P, Chien R et al. The KAPA HyperCap Design Share NHL Panel enables highly sensitive,
longitudinal detection of non-Hodgkin lymphoma circulating tumor DNA. Roche white paper. MC-US-13745.
*7. Herrera et al. Risk Profiling of Patients with Previously Untreated Diffuse Large B-Cell Lymphoma (DLBCL) By Measuring Circulating Tumor DNA (ctDNA): Results from the POLARIX Study. Blood 2022; 140 (supplement 1): 1297-1300. https://pubmed.ncbi.nlm.nih.gov/35086141/
*8.Newman AM, Lovejoy AF, Klass DM, et al. Integrated digital error suppression for improved detection of circulating tumor DNA.
Nature Biotechnol 2016;34(547 – 555).
doi: 10.1038/nbt.3520.
*9.Alkodsi A, Meriranta L, Pasenen A, Sirpa Leppä. ctDNAtools: An R package to work with sequencing data of circulating tumor DNA. bioRxiv 2020.01.27.912790.
doi: 10.1101/2020.01.27.912790.